Tradução de trecho do artigo:
New insights on the antiviral effects of chloroquine against coronavirus: what to expect for COVID-19? - Christian A. Devaux, Jean-Marc Rolain, Philippe Colson, Didier Raoult.
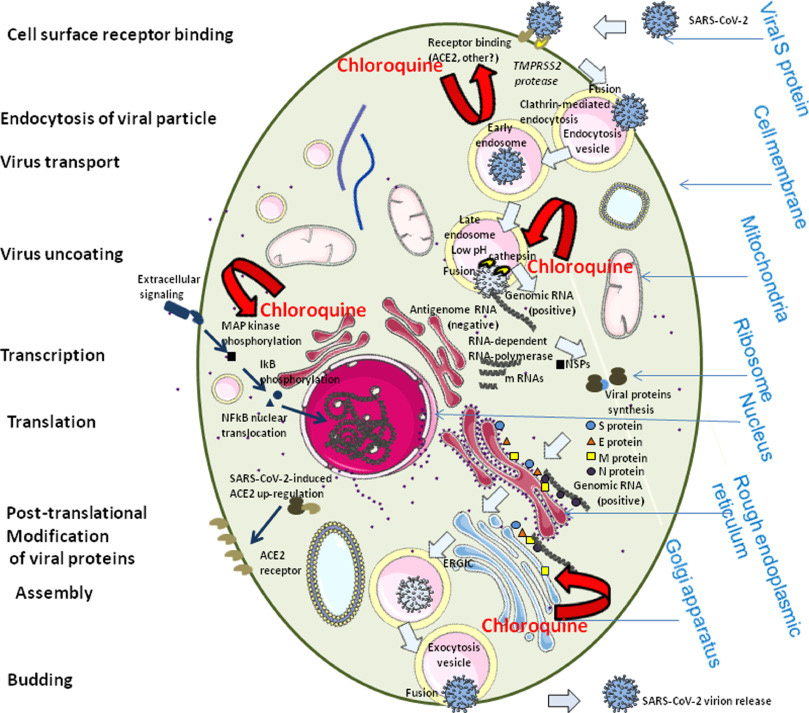
Representação esquemática dos possíveis efeitos da cloroquina no ciclo de replicação do coronavírus de síndrome respiratória sever aguda 2 (SARS-CoV-2). O SARS-CoV-2, como outros coronavírus humanos, têm três proteínas de envelope, a proteína Spike (S) (180 - 220 kDa), a proteína membrana (M) (25 - 35 kDa) e a proteína envelope (E) (10 - 12 kDa), que são necessárias para a entrada do vírion (partícula viral) infeccioso na célula alvo. O vírion também contém o nucleocapsídio (N), capaz de se ligar ao genoma viral RNA e a proteína nsp3, um componente chave para o complexo replicase. A glicoproteína S determina o tropismo celular do hospedeiro. Há uma indicação que o SARS-CoV-2 se liga à enzima conversora de angiotensina 2 (ACE2) expressa nos pneumócitos. É previsto que a ligação ao ACE2 aciona as mudanças conformacionais na glicoproteína S, permitindo a clivagem pela protease transmembrana TMPRSS2 da proteína S e a liberação dos fragmentos S no sobrenadante celular que inibe a neutralização viral por anticorpos. O vírus é então transportado para dentro da célula através dos endossomos iniciais e tardios onde a protease hospedeira catepsina L continua a clivagem da proteína S em pH baixo, levando à fusão do envelope viral e membrana fosfolipídica dos endossomos, resultando na liberação do genoma viral no citoplasma celular. A replicação então inicia e a fita positiva do genoma viral RNA é transcrito numa fita negativa de RNA que é usada como molde para a síntese de mRNA viral. A síntese da fita negativa de RNA tem auge mais cedo e declina mais rápido do que a síntese da fita positiva. Célula infectadas contém entre 10 e 100 vezes mais fitas positivas do que negativas. A maquinaria ribossomal das células infectadas são desviadas à favor do vírus, que então sintetiza suas proteínas não estruturais (NSP) que montam no complexo replicase-transcriptase para favorecer a síntese no subgenoma viral mRNA. Seguindo a replicação, as proteínas do envelope são traduzidas e inseridas no retículo endoplasmático e então movidas para o complexo de Golgi. O genoma viral RNA é levado para dentro do nucleocapsídio e as proteínas envelope são incorporadas durante a etapa de brotamento para formar vírions maduros. A proteína M, que se localiza na rede trans-Golgi, tem parte essencial durante a montagem viral ao interagir com as outras proteínas do vírus. Após a montagem, as novas partículas virais recém formadas são transportadas para a superfície celular em vesículas e são liberadas por exocitose. É possível que a cloroquina interfira com a glicolisação do receptor ACE2, portanto, previnindo a ligação do SARS-CoV-2 às células alvo. A cloroquina pode também, possivelmente, limitar a biossíntese de ácidos siálicos que podem ser necessários para a ligação do coronavírus à superfície celular. Se a ligação de partículas virais são efetivas, a cloroquina pode modular a acidificação dos endossomos e assim inibindo a formação do autofagossomo. Pela redução da ativação da proteína quinase ativada por mitógeno (MAPK), a cloroquina também pode vir a inibir a replicação viral. Ainda, a cloroquina pode alterar a maturação da proteína M e interferir com a montagem e brotamento do vírion.
Schematic representation of the possible effects of chloroquine on the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) replication cycle. SARS-CoV2, like other human coronaviruses, harbours three envelope proteins, the spike (S) protein (180–220 kDa), the membrane (M) protein (25–35 kDa) and the envelope (E) protein (10–12 kDa), which are required for entry of infectious virions into target cells. The virion also contains the nucleocapsid (N), capable of binding to viral genomic RNA, and nsp3, a key component of the replicase complex. A subset of betacoronaviruses use a hemagglutinin-esterase (65 kDa) that binds sialic acids at the surface of glycoproteins. The S glycoprotein determines the host tropism. There is indication that SARS-CoV-2 binds to angiotensin-converting enzyme 2 (ACE2) expressed on pneumocytes [85,99]. Binding to ACE2 is expected to trigger conformational changes in the S glycoprotein allowing cleavage by the transmembrane protease TMPRSS2 of the S protein and the release of S fragments into the cellular supernatant that inhibit virus neutralisation by antibodies [100]. The virus is then transported into the cell through the early and late endosomes where the host protease cathepsin L further cleaves the S protein at low pH, leading to fusion of the viral envelope and phospholipidic membrane of the endosomes resulting in release of the viral genome into the cell cytoplasm. Replication then starts and the positive-strand viral genomic RNA is transcribed into a negative RNA strand that is used as a template for the synthesis of viral mRNA. Synthesis of the negative RNA strand peaks earlier and falls faster than synthesis of the positive strand. Infected cells contain between 10 and 100 times more positive strands than negative strands. The ribosome machinery of the infected cells is diverted in favour of the virus, which then synthesises its non-structural proteins (NSPs) that assemble into the replicase-transcriptase complex to favour viral subgenomic mRNA synthesis (see the review by Fehr and Perlman for details [101]). Following replication, the envelope proteins are translated and inserted into the endoplasmic reticulum and then move to the Golgi compartment. Viral genomic RNA is packaged into the nucleocapsid and then envelope proteins are incorporated during the budding step to form mature virions. The M protein, which localises to the trans-Golgi network, plays an essential role during viral assembly by interacting with the other proteins of the virus. Following assembly, the newly formed viral particles are transported to the cell surface in vesicles and are released by exocytosis. It is possible that chloroquine interferes with ACE2 receptor glycosylation, thus preventing SARS-CoV-2 binding to target cells. Chloroquine could also possibly limit the biosynthesis of sialic acids that may be required for cell surface binding of SARS-CoV-2. If binding of some viral particles is achieved, chloroquine may modulate the acidification of endosomes thereby inhibiting formation of the autophagosome. Through reduction of cellular mitogen-activated protein (MAP) kinase activation, chloroquine may also inhibit virus replication. Moreover, chloroquine could alter M protein maturation and interfere with virion assembly and budding.


